자유게시판
Home > 정보교류센터 > 자유게시판| SARS-CoV-2 전사체 발현의 모든 가능성을 최초로 분석한 논문 The architecture of SARS-CoV-2 transcriptome |
|
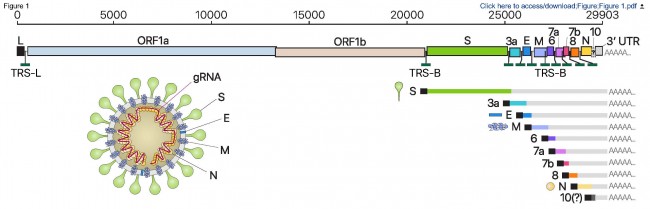
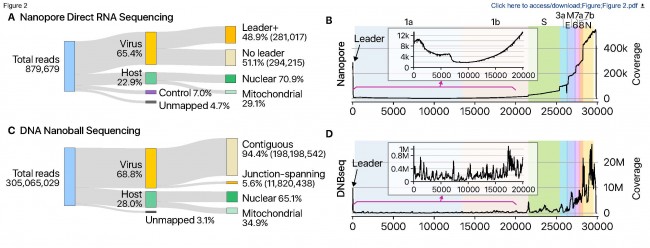
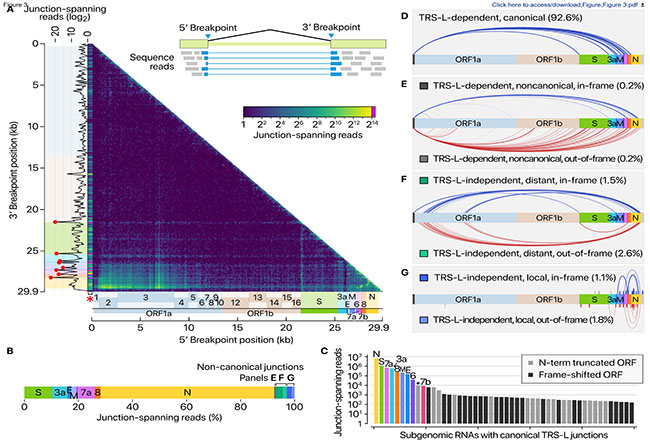
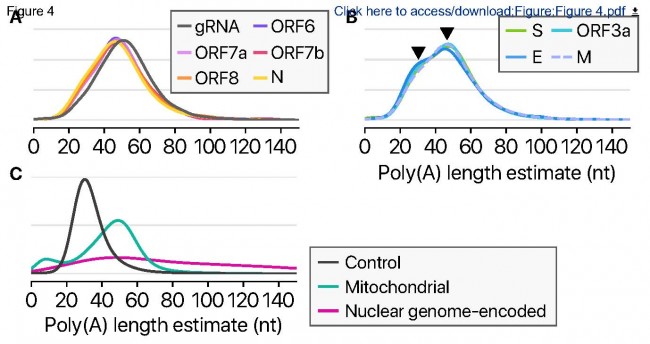
SARS-CoV-2 전사체 발현의 모든 가능성을 최초로 분석한 서울대학교 장혜식 교수님의 논문이 공동저자로 IBS RNA 연구단장이신 김빛내리 교수님과 한국 질병관리본부 관계자 분들도 포함되어있네요. 옥스포드 나노포어 시퀀싱 플랫폼의 롱리드 방법을 활용해 코로나바이러스가 숙주 세포 내에서 만들어 내는 수백 만~수천 만 개의 서브 지노믹 RNA (sgRNA)의 모든 가능성을 정성/정량 분석한 첫 연구 결과입니다. 게다가 SARS-CoV-2 게놈 내 발현된 전사체에서 RNA modifications이 일어나는 패턴까지도 분석해 코로나바이러스의 숙주 세포 면역 체계를 회피하는 방식에 대해서도 이 연구를 통해 이해할 수 있습니다. 논문에서 차세대유전체해독(NGS) 기술로 엄청나게 딥 시퀀싱(deep sequencing)을 했음에도 ORF10이 발현이 거의 안 되는 것이 너무 특이하네요. 정말 대단한 것은 이 연구에 활용된 모든 데이터와 소스코드까지 모두 공유하셨네요~ 또한 최근에는 UCSC 지놈 브라우저에서 시각적으로 코로나바이러스의 전사체 구조까지 들여 다 볼 수도 있게 전 세계 연구자들에게 공유해주고 있습니다. 지금 전 세계 사람들을 아프게하고 아까운 생명을 빼앗아가는 이 코로나바이러스를 우리가 좀 더 잘 이해할 수 있게 되었고 그것도 한국에서 이런 훌륭한 연구 결과가 나올 수 있었다는 것에 너무 기분이 좋습니다.
논문 링크: The architecture of SARS-CoV-2 transcriptome 시퀀싱 데이터: https://osf.io/8f6n9/ 소스코드: https://github.com/hyeshik/sars-cov-2-transcriptome UCSC 지놈 브라우저: http://genome.ucsc.edu/cgi-bin/hgTracks?db=wuhCor1&lastVirtModeType=default&lastVirtModeExtraState=&virtModeType=default&virtMode=0&nonVirtPosition=&position=NC_045512v2%3A27573%2D29903&hgsid=821872279_DysINWmGHHFQE2gsanIhyDjyy1L5
|