동향분석
Home > 뉴스브리핑센터 > 동향분석| 한약재 추출물 (TCM-33)에 의한 Mouse hepatitis virus A-59 의 증식 억제와 기작 규명 (아주대 의대 김경민 교수팀, 2011) |
|
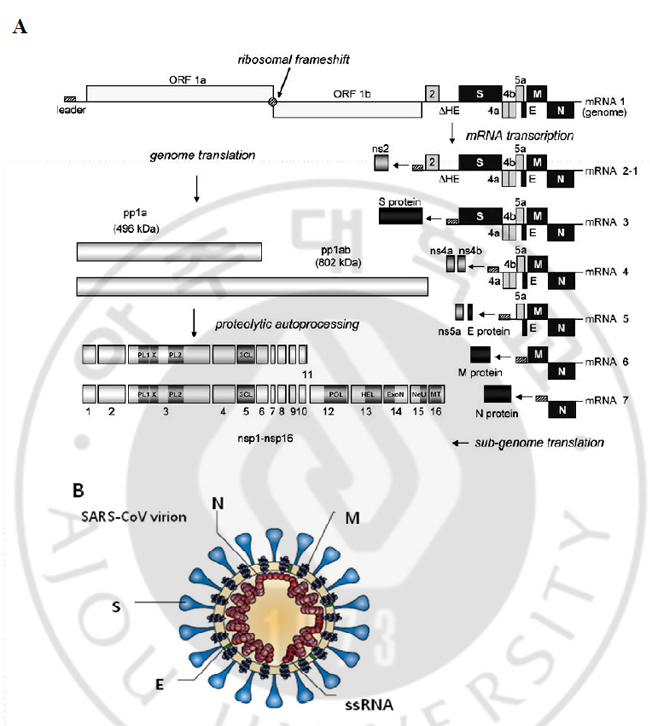
한약재 추출물 (TCM-33)에 의한 Mouse hepatitis virus A-59 의 증식 억제와 기작 규명 어은영, 김경민 (아주대학교 대학원 의생명과학과/분자의학전공) 국문 요약 현재까지의 연구에서 승마 (cimicifuga fhizoma), 고련피 (meliae cortex), 황련 (coptidis rhizoma), 황백 (phellodendeon cortex), 고삼 (sophorae radix), 오가피 (acanthopanacis cortex), 지유 (sanguisorbae radix), 사상자 (torilis fructus)가 항코로나바이러스 효과가 있다고 보고되었다. 본 연구에서는 TCM-33이 in vitro에서 항코로나바이러스 효과가 있는지 알아보고자 하였다. TCM-33는 마우스 간염 바이러스 (mouse hepatitis virus-A59, MHV-A59)의 생산을 억제하였으며, TCM-33의 농도에 따라 MHV-A59의 증식이 억제되었다. 이때, 바이러스의 mRNA와 단백질 합성이 현저히 감소하였다. TCM-33의 EC50(effective concentration)과 CC50(cytotoxic concentration)는 각각 4.8±1.4 μg/ml, 519.7±50.3 μg/ml이고, SI (selectirity index)는 107.6 이었다. TCM-33의 항코로나바이러스 효과의 기작을 알아보기 위하여 TCM-33이 MHV-A59 증식 단계 중 어느 단계에서 효과를 나타내는지 관찰하였다. TCM-33이 바이러스 진입에는 부분적으로 영향을 미칠 것으로 추정되지만, 주요 기작은 아니라고 생각되었다. 또한 TCM-33은 바이러스의 음성가닥 RNA 합성을 현저하게 감소시켰으나 세포의 단백질 합성은 감소하지 않았다. 따라서 바이러스의 초기 단백질 합성에는 영향이 거의 없을 것으로 추정하였다. 즉, TCM-33이 아마도 MHV-A59 증식에서 초기 단계에 억제 효과를 보이는 것으로 생각되며, 비구조 protein processing 과/또는 RNA 합성일 것으로 추정하였다. keywords : 한약재 추출물, mouse hepatitis virus, minus-strand RNA 합성 Ⅰ. 서론 A. 코로나바이러스 코로나바이러스는 60년 전에 처음으로 밝혀졌지만, 2002~2003 년에 발발한 중증 급성 호흡기 증후군 (severe acute respiratory syndrome, SARS)이 SARS 코로나바이러스에 의해 유발된다는 것이 알려지면서 일반인들에게 널리 알려지게 되었다. 코로나바이러스는 척추동물에서 조류에 이르기까지 사람과 동물에게 모두 감염되고, 위장염, 간염, 호흡계 질병, 신경계 질병 등 다양한 질병을 일으킨다 (Weiss and Navas-Martin, 2005). 코로나바이어스 중 MHV (mouse hepatitis virus)는 세포배양이 용이하여 가장 많은 연구가 이루어진 바이러스이다. 또한 MHV는 SARS-CoV 와 분류상 같은 그룹에 속하고, 항원성 및 염기서열이 높은 유사성 (homology)를 가진다. 코로나바이러스는 coronaviridae 과에 속하며, 구형의 외피를 가진 바이러스이며, 양성 단일 가닥의 RNA 유전체를 가진다. 유전체의 크기는 대략 27~32kb 이며, 유전체의 2/3 를 차지하는 Gene 1 (ORF1a/ab)은 증식에 필요한 여러 비구조 단백질 (non-structural protein)을 암호하고 있고, 나머지 유전체의 1/3 은 구조 단백질 (structural protein)을 암호하고있다: spike (S), envelope(E), membrane (M), nucleocapside (N) 일부 group 2 코로나바이러스 중에는 haemagglutinin-esterase (HE)를 암호하고 있는 것도 있다 (Perlman and Netland, 2009) (Fig. 1).
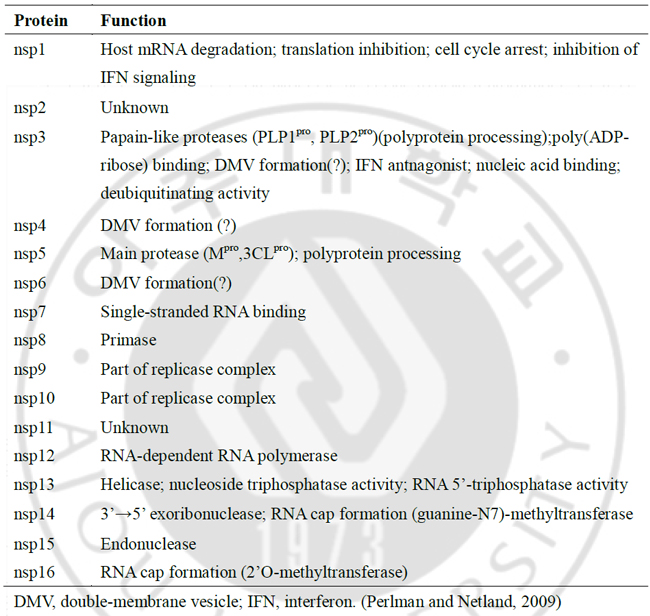
Fig. 1. Organization and expression of coronavirus genome and virion. (A) Schematic diagram of MHV-A59. Approximately the first two-thirds of the 32kb, positive-sense RNA genome encodes a large polyprotein (ORF1a/b) that is proteolytically cleaved to generated 16 non-structural proteins. The 3’-end one-third of the genome encodes four structural proteins-spike (S), membrane (M), envelope (E), nucleocapsid (N) and haemagglutitinesterase (HE). (B) Schematic diagram of the coronavirus virion (Stadler et al., 2003; Sawicki et al., 2005). B. 비구조 단백질과 구조 단백질 코로나바이러스는 15-16개의 비구조 단백질 (non -structural protein, nsp)를 암호하고 있다. 비구조 단백질들은 증식에 관여한다고 알려져 있지만, 몇몇 비 구조 단백질의 기능만이 밝혀진 상태이고, 모든 비구조 단백질의 기능은 아직까지 완전히 밝혀지지 않았다. nsp 3는 papain like proteases 기능을 가지고 있어서 protein processing에 관여한다고 알려져 있고, protease domain에 deubiquitylating 활성이 있다. nsp 5는 3CLpro (chymotrypsin-like protease) 또는 Mpro (main protease)라고 알려진 protease 활성을 가지고 있다. nsp 1는 RNA-dependent RNA polymerase (RdRp)로서 작용을 하고, nsp 13은 helicase로 작용을 한다. nsp 8 은 RNA 증식에 필요한 RNA primer 을 합성하는 primase 로서 작용을 하고 (Imbert et al., 2006), nsp 7, nsp8, nsp9, nsp10은 subgenomic mRNA 와 genomic RNA 증식에 관여한다고 알려져 있다 (Deming et al., 2007). nsp7 과 nsp 8은 hexadecameric 구조이며, RNA에 결합할 수 있는 활성을 가지고 있다 (Zhai et al., 2005). nsp 14는 3’→5’ exonuclease 기능을 가지고 있으며, (guanine-N7)-methyl transferase (N7-MTase) 활성을 가지고 있어서 RNA cap 형성에 관여한다고 알려져 있다 (Chen et al., 2009). nsp 15는 uridylate-specific endoribonuclease (Nendo U) 활성을 가지고 있어서 RNA cleavage에 관여하지만 바이러스 증식에서의 영향은 알려지지 않았다 (Ivanov et al., 2004). nsp 16은 S-adenosyl-L- methionie-dependent RNA (nucleoside-2’O)-methyltransferase (2’O-MTase)라는 nsp 14와 유사한 기능을 하며, RNA cap 형성에 관여한다고 알려져 있다 (Decroly et al., 2008) (Table. 1). Table 1. Coronavirus non-structural proteins and their functions.
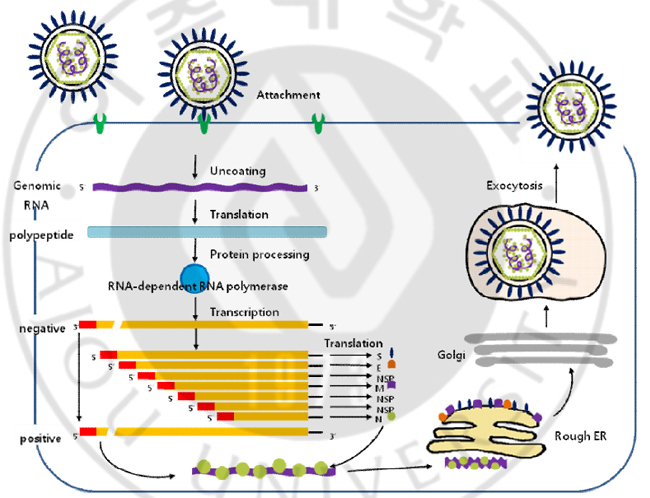
코로나바이러스의 바이러스 입자에는 4개 또는 5개의 구조 단백질이 있다: spike (S), membrane (M), haemagglutinin-esterase (HE), nucleocapsid (N), envelope (E). S (spike) 단백질은 바이러스의 외피에 spike를 구성하며, 분자량은 180kDa이다. S 단백질은 N-말단에 signal sequence, C-말단에 소수성 transmembrane domain, 다수의 potential N-linked glycosylation site 가 있다 (Stern and Sefton, 1982). S 단백질은 표적세포상에 바이러스를 인식하는 수용체에 코로나바이러스를 부착시키는데 관여한다. M (membrane) 단백질은 막을 3 번 통과하는 당 단백질이며, 분자량이 20~30kDa이다. M 단백질은 세포질 쪽에 큰 카르복실 말단을 가지고 있고, 세포질에서 발현되어 소포체와 cis-Golgi에 위치하기 때문에 코로나바이러스의 조립에 있어서 주요한 인자라고 추정된다고 보고되었다 (Tooze et al., 1984). N (nucleocapsid) 단백질은 virion RNA 와 결합하며, 분자량이 50~60kDa인 인산화 단백질 (phosphoprotein)이다. N 단백질의 인산화는 전사 후에 일어나며, 이런 인산화는 N 단백질과 virion RNA 사이의 결합을 강하게 해줄 것이라고 보고되었다 (Stohlman and Lai, 1979). 또한 N 단백질은 다른 N, M 단백질과 결합하며, interferon (IFN) antagonist 로서 작용을 한다. E (envelope) 단백질은 작은 막 단백질이며, 형태 형성, 조립, 방출에 관여한다. E 단백질은 ion channel 활성을 가지고 있어서, 바이러스 증식에 관여한다고 알려져 있다 (Wilson et al., 2004; Madan et al., 2005). HE (haemagglutitin-esterase) 단백질은 분자량이 65kDa이고, virion의 작은 spike를 구성한다. Bovine coronavirus (BCoV), Turkey coronavirus (TCoV), Human coronavirus (HCoV), Mouse hepatitis virus (MHV)에서는 존재하지만, IBV 와 TGEV에는 존재하지 않는다. BCoV의 HE 단백질은 haemagglutinating 과 esterase 활성을 가진다. 그러나 바이러스 증식에는 필요하지 않지만 숙주세포에 감염할 때 중요한 역할을 할 것이라고 보고되었다 (Lai, 1990). C. 코로나바이러스 증식 코로나바이러스는 세포질에서 증식을 하며, 코로나바이러스 중 MHV는 세포배양이 용이하여 가장 많은 연구가 이루어진 바이러스이다. MHV의 증식은 자신의 S 단백질과 세포 표면에 있는 CEACAM-1이라는 수용체와 상호작용하여 세포 표면에 부착하면서 시작된다. 세포 안으로 들어간 MHV는 자신의 virion을 벗고, genomic RNA를 세포질에 방출한다. 방출된 genomic RNA는 양성 단일 가닥 RNA이기 때문에 바로 번역이 일어나서 polypeptide를 형성한다. MHV-A59의 gene 1은 genome 의 2/3 를 차지하고, ORF1a 와 ORF1b 가 overlapping 되어 있으며 (Bredenbeek et al., 1990), fameshift에 의하여 ORF1ab 가 번역되어 800kDa의 거대한 polypeptid (ORF1ab)가 합성된다. ORF1a에는 3가지 protease domain이 있고: cysteine proteases인 PLP1과 PLP2, poliovirus 3C-like protease, ORF1b에는 RNA-dependent RNA polymerase (RdRp)가 있다. 이 polypeptide (ORF1a/ab)는 바이러스 유전체에 암호화되어있는 3CLpro 또는 추가적으로 PLP1pro/PLP2pro에 의하여 autoproteolytic processing이라는 과정에 의하여 16 개의 비구조 단백질이 만들어진다 (Masters, 2006). 여러 증식에 관여하는 바이러스 단백질과 숙주세포의 단백질들이 조립되어 하나의 특정 replicase-transcriptase complex를 이루며 (Sawicki et al., 2005), 이 복합체에 의하여 genomic RNA로부터 전체 genomic 크기의 음성가닥 RNA와 sub-genomic 크기의 음성가닥 RNA를 합성한다 (Sawicki and Sawicki, 1986; Sawicki et al., 2001). 이 음성가닥 RNA를 주형으로 하여 genomic RNA와 subgenomic mRNA를 합성한다. Subgenomic mRNA에서 각각 N, M, E, S, HE 단백질들이 합성되고, N 단백질이 새로이 합성된 genomic RNA와 세포질에서 조립되어 helical nucleocapsid를 형성한다. 소포체와 골지체에서 M과 E 단백질이 nucleocapsid 와 결합하고, S와 HE 단백질도 소포체에서 골지체로 이동하는 동안에 결합하여 완전한 virion을 형성하게 된다. 만들어진 virion은 숙주세포에서 방출된다 (Fig. 2).
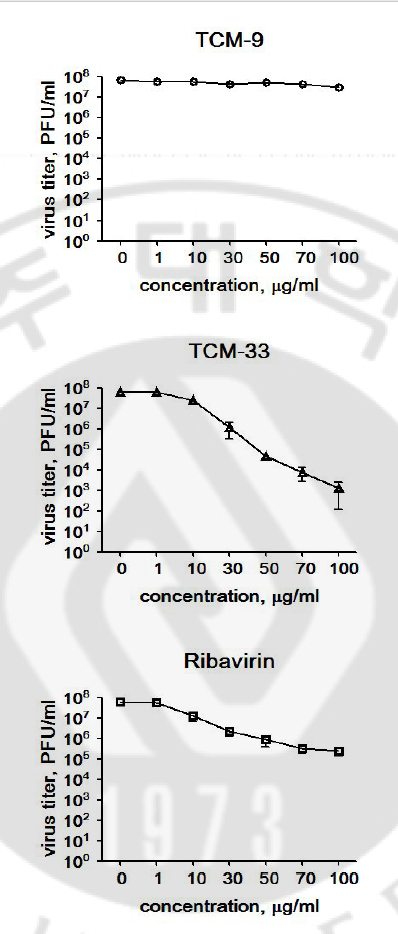
Fig. 2. Replicaiton cycle of MHV-A59. The life cycle of MHV-A59 starts when the spike (S) protein interacts with a receptor. The RNA genome is then release into the cytoplasm where replication take place. The host translation machinery translates the overlapping ORF1a and ORF1b by a ribosomal frame-shifting mechanism to produce a single polyprotein. Cleavage by virally encoded proteinases yields the components that are necessary to assemble the viral replication complex, which synthesizes full-length negative-strand RNA. In addition, a discontinuous transcription strategy during negative-strand synthesis produces a nested set of sub-genomic negative-sense RNAs. These discontinuously synthesized minus strands then act as templates for the synthesis of positive-sense mRNAs. An alternative hypothesis proposes that these mRNA molecules are generated by discontinuous transcription during positive-strand synthesis. Nucleocapsid (N) protein and genomic RNA assemble in the cytoplasm to form the helical nucleocapsid. This core structure acquires its envelope by budding through intracellular membranes between the endoplasmic reticulum (ER) and the golgi apparatus. The membrane (M), envelope (E) and S protein are transported through the ER to the budding compartment, where the nucleocapsid probably interacts with the M protein to trigger assembly. Finally, the virus is released from the host cell by fusion of virion-containinig vesicles with the plasma membrane. 2002~2003년의 SARS의 창궐 이전에는 코로나바이러스가 동물에서 다양한 질병을 유발하고, 사람에서는 일반적인 감기만을 유발한다고 알려져 있었다. 그러나 SARS의 원인이 SARS 코로나바이러스 (SARS-CoV)라고 밝혀지면서 코로나바이러스에 대한 활발한 연구가 이루어지고 있다. 또한 SARS 이후로 몇 가지 항 SARS 코로나바이러스제 연구가 진행되어 왔지만, 효과적인 항바이러스제는 아직까지 보고된 바가 없다. 합성 리보 핵산의 일종인 ribavirin 이 SARS에 대한 항바이러스제로서 사용되고 있지만, 아직까지 확실한 효과는 불분명하다. 그러나 in vitro에서는 SARS에 대해 약간의 항바이러스 효과를 보였다 (Groneberg et al., 2005). Glycyrrhizin, pyridine N-oxidederivatives, ATPase, helicase inhibitor, 4-aminoquinoline 이 in vitro에서 SARS-CoV의 증식을 억제한다고 밝혀졌다 (Cinatl et al., 2003; Keyaerts et al., 2004; Cinatl et al., 2005; Tanner et al., 2005; Balzarini et al., 2006). 최근에 한약재 추출물인 승마 (cimicifuga fhizoma), 고련피 (meliae cortex), 황련 (coptidis rhizoma), 황백 (phellodendeon cortex), 고삼 (sophorae radix), 오가피 (acanthopanacis cortex), 지유 (sanguisorbae radix), 사상자 (torilis fructus)가 in vitro에서 코로나바이러스 증식을 억제한다고 보고하였다 (Kim et al., 2008; Kim et al., 2010). 본 연구에서는 새로운 한약재 추출물 (TCM-33 extract)이 코로나바이러스에 대한 항코로나바이러스 효과가 있는지를 알아보고, 항코로나바이러스 효과가 있다면, 한약재 추출물이 바이러스 증식 과정 중 어느 단계에서 효과를 나타내는지를 규명하고자 하였다. Ⅱ. 재료 및 방법 A. 세포 배양과 바이러스 감염 Delayed brain tumor (DBT) 세포를 MEM (minimum essential medium, Gibco BRL, Grand Island, NY) 8% bovine serum (Gibco BRL, Grand Island), 10% tryptose phosphate broth (TPB) (Sigma Aldrich), penicillin 과 streptomysin (Gibco BRL, Grand Island) 배지에 배양하였다. 바이러스는 mouse hepatitis virus-A59 (MHV-A59) strain를 사용하였다. MHV-A59은 DBT 세포에서 증식하였고, 바이러스 titer는 plaque assay를 통하여 plaque formation unit (PFU)로 나타내었다. 바이러스 감염은 DBT 세포에 2MOI (multiplicity of infection)의 MHV-A59을 37℃에서 한 시간 동안 감염시켰다. 접종원을 제거한 후, 배지 (2% FBS, 10% TPB, 1% PS, 87% MEM)를 첨가하고 37℃에서 배양하였다. B. 한약재 추출물 실험에 사용한 한약재 추출물은 원광대로부터 받았다. 각각의 식물을 메탄올로 추출한 뒤 여과를 하고, 각각의 용매를 증발시켰다. 그 후 남은 한약재 및 천연 약물 추출물을 DMSO (dimethyl sulfoxide) (Sigma Aldrich)에 녹였다. C. Plaque assay 약재에 의한 바이러스의 titer의 변화를 알아보기 위하여, 바이러스를 세포에 감염시킬 때, 약재를 같이 처리하여 37℃에서 12시간 동안 배양한 후, 바이러스가 배출된 세포 배양액를 수확하였다. 수확한 바이러스를 MEM으로 10배씩 순차적으로 희석하였고, 희석한 바이러스를 DBT 세포가 90% 정도 자란 6 well plate에 처리한 후, 37℃에서 1시간 동안 배양하였다. 0.9% noble agar (USB, 10907)가 포함된 배지를 넣어준 후, noble agar가 굳으면 뒤집어서 37℃에서 48시간 동안 배양하였다. 0.25% neutral red를 이용하여 염색을 한 다음 plaque 형성을 보았다. D. MTT assay MTT(3-[4,5-dimethylththiaxol-2-yl]-2,5-diphenyltetrazolium bromide) assay를 통해서 TCM-33 추출물이 DBT 세포에 세포 독성 효과를 보이는지를 알아보고자 하였다. DBT 세포를 96-well microplates에 완전히 부착시킨 후 각각의 천연 약재 추출물을 알맞은 농도로 희석한 후 12시간 동안 37℃에서 배양시켰다. 세포 독성을 알아보기 의해 1.5 mg/ml의 MTT를 넣은 후 3시간 동안 배양한 후 Fomazen을 DMSO에 녹여 ELISA plate reader로 595 nm에서 측정하였다. E. 세포 내 RNA 추출과 Northern blot 바이러스가 감염된 세포에서 바이러스의 mRNA를 확인하기 위하여, MHVA59를 5시간 30분 동안 배양시킨 세포로부터 세포 내 RNA를 추출하였다. 간단히 설명하면, 세포를 PBS를 이용하여 부유시켰고, 세포를 침전시켜서 lysis buffer (100mM NaCl, 10mM Tris-HCl (pH 7.5), 1mM EDTA, 0.5% NP-40)로 용해하였다. 용해물 (lysates)에 2X protease K buffer(0.3M NaCl, 0.2M Tris-HCl (pH 7.5), 25mM EDTA, 2% SDS)와 proteinase K (100㎍/ml)를 37℃에서 30분간 처리한 뒤, phenol 과 chloroform 추출방법을 이용하여 RNA를 추출하였다. 3 ㎍의 RNA를 1% agarose gel에 전기영동한 뒤, nylon membrane에 이동시킨 후, MHV의 gene 7에 특이적인 32P-labeled random primed probe를 사용하여 Northern blot를 수행하였다. F. SDS-PAGE와 Western blot MHV-A59을 DBT 세포에 5시간 30분 동안 배양 시킨 후, 세포에 lysis buffer (1% Triton X-100, 0.5% sodium deoxycholate, 0.1% sodium dodecyl sulfate (SDS), 100mM phenylmethylsulfonyl fluoride (PMSF))를 처리하여 세포를 용해시켰고, 용해물에 19G 주사기로 5번 통과시켜 준 뒤에 세포를 침전시킨 후 용해물을 준비하였다. 용해물에 2X sample buffer (0.5M Tris-HCl (pH 6.8), 10% SDS, 10% glycerol, 0.2 % bromophenol, 4% β-mercaptoethanol)를 첨가하여 100℃에서 6분 동안 처리 한 후에, 10% SDS-PAGE 하고, polyvinylidene fluoride (PVDF) membrane으로 이동시켰다. 각각 MHV의 N 단백질에 대한 단일클론 항체 (monoclonal antibody)는 5% skim milk in PBS.T에 1:2000으로 희석하여 사용하였고, M 단백질에 대한 단일클론 항체 (monoclonal antibody)는 원액을 사용하였고, α-tubulin (Anti-α-tubulin mouse mAb (DM1A): CALBIOCHEM, USA, Cat. #cp06)에 대한 항체는 5% skim milk in PBS.T에 1:1000으로 희석하여 1차 항체로 사용하였고, polyclonal rabbit anti-mouse immunoglobulin/HRP, (Dako Cytomation Denmark, Cat. P0260))을 5% skim milk in PBS.T에 1:2000으로 희석하여 2차 항체로 사용하였다. N과 M의 단일클론 항체는 JO Fleming (university of Wisconsin school of medicine and public health, Madison, WI, USA)으로부터 받았다. G. 전체 RNA 추출과 RT-PCR DBT 세포에 2MOI로 MHV-A59를 1시간 동안 감염시킨 다음, 3시간 동안 배양 후, DBT 세포를 RNA iso plus (TaKaRa, Cat. #9108)를 사용하여 전체 RNA를 추출하였다. 3 ㎍의 Total RNA를 RevertAidTM First strand cDNA synthesis kit (Fermentas)을 사용하여 cDNA를 합성하였다. 음성가닥 RNA을 관찰하기 위하여 사용한 primer (435-456nt): 5’-ATA ATC CGT GAT ATT TTT GAT G-3’, 양성가닥 RNA 을 관찰하기 위하여 사용한 primer(744-766nt): 5’-TGC AAC CAC TGC TTC GCA TAC TC-3’, actin을 관찰하기 위하여 oligo(dT)를 사용하여 reverse transcription (RT)를 하여 cDNA를 합성하였다. 2 ㎕의 cDNA를 사용하여 음성가닥 RNA와 양성가닥 RNA는 forward: 5’-ATA ATC CGT GAT ATT TTT GAT G-3’, reverse: 5’-TGC AAC CAC TGC TTC GCA TAC TC-3’을 사용하여 증폭하였고, actin은 forward: 5’- CAG GTC ATC ACC ATT GGC AAT GAG-3’, reverse: 5’-CAG CAC TGT GTT GGC GTA CAG GTC-3’을 사용하여 증폭하였다. PCR 반응은 94℃에서 40초 52℃에서 40초 72℃에서 1분 동안 총 35사이클 (cycle) 하였다. H. Virion 분리 및 RNA 추출 MHV-A59을 DBT 세포에 12시간 동안 배양한 뒤, 바이러스가 방출된 세포 배양액을 수확하였다. 수확한 바이러스 부유액으로부터 sucrose gradient 원심분리 방법을 이용하여 virion을 분리하였다. 간단히 설명하면, 20% sucrose 위에 바이러스 부유액을 올리고 4℃에서 26,000 rpm으로 3시간 동안 원심분리하였다. 상층액을 버리고 침전물 (pellet)을 NTE buffer (0.1M NaCl, 10mM Tris-HCl (pH 7.5), 1mM EDTA)로 부유시켰다. 이 부유액을 바닥부터 60%, 50%, 30%, 20%의 순서로 만든 sucrose 위에 올려놓고 4℃에서 26,000 rpm으로 3시간 동안 원심분리하였다. 30~50% sucrose 사이의 층을 취한 후 NTE buffer로 희석시켜주었다. 20% sucrose 위에 이 부유액을 올리고 4℃에서 26,000 rpm으로 3시간 동안 원심분리하였다. 침전물을 T.E buffer (10mM Tris-HCl (pH 7.5), 1mM EDTA)로 부유시킨 후, 2X protease K buffer (0.3M NaCl, 0.2M Tris-HCl (pH 7.5), 25mM EDTA, 2% SDS), protease K (100㎍/ml)를 넣고 37℃에서 30분 동안 반응시킨 후, phenol과 chloroform 추출 방법을 통하여 virion RNA을 추출하였다. I. 세포의 단백질 합성 조사 바이러스를 세포에 감염 시켰을 때와 약재를 처리하였을 때, 세포의 단백질 합성이 영향을 받는지 알아보기 위하여, MHV-A59를 DBT 세포에 4℃에서 1시간 동안 처리하여 바이러스가 숙주세포의 막에만 부착되고 세포 내로 들어가는 것을 억제하였다. MEM 배지 (2% FBS, 1X TPB, 1% PS)를 첨가할 때, 약재를 50㎍/ml로 처리하여 37℃에서 2시간 10분 동안 배양한 후, methionine free media를 넣고 37℃에서 30분 동안 처리하였다. L-35S methionine (NEG-709)를 100 μci/ml로 37℃에서 20분 동안 처리한 후, 단백질을 lysis buffer를 처리하여 용해시켰고, 19G 주사기로 5번 통과하였다. 13,000 rpm에서 30초간 원심분리하여 상층액을 분리하였다. 용해물에 2X sample buffer를 처리하여 100℃에서 7분 동안 처리한 후, 10% SDS-PAGE 하였다. Destaining solution (45% methanol, 10% glacial acetic acid)을 넣고 25℃에서 1시간 동안 처리한 후, Enhance solution (1M sodium salicylate)을 25℃에서 1시간 동안 처리하였다. Gel을 80℃에서 90분 동안 건조시켜 주고, 자가방사도법 (autoradiographic)으로 확인하였다. 약재에 의한 전체 단백질 합성 수준을 조사하기 위하여, MHV-A59를 DBT 세포에 4℃에서 1시간 동안 처리하였다. MEM media (2% FBS, 1X TPB, 1% PS)를 첨가할 때, 약재를 동시에 처리한 후 37℃에서 3시간 동안 처리하였다. 감염된 세포에 lysis buffer를 처리하여 용해시킨 후, 19G 주사기로 5번 통과하였다. 13,000 rpm으로 30초간 원심분리하여 상층액을 분리하였다. 용해물에 2X sample buffer를 처리하여 100℃에서 7분 동안 처리한 후, 10% SDS-PAGE 하였다. Coomassie staining solution (0.25% coomassie brilliant blue R-250 (v/v), 10% glacial acetic acid, 90% methanol)을 25℃에서 1시간 동안 처리한 후, destaining solution (45% methanol, 10% glacial acetic acid)을 25℃에서 처리하였다. J. Proteinase 억제제 (inhibitor) 실험 Proteinase 와 proteasome 억제제를 처리하였을 때, 음성가닥 RNA 합성을 알아보기 위하여, DBT 세포에 2MOI의 MHV-A59을 4℃에서 1시간 동안 처리한 후, proteinase와 proteasome 억제제를 Antipain은 100μM, chymostatin은 50 μM, E-64는 1μM, E-64는 100㎍/ml, epoxomicin은 1μM, MG132는 10μM으로 처리하고 37℃에서 3시간 동안 배양하였다. 전체 RNA을 추출한 뒤, 음성가닥 RNA의 수준을 알아보기 위하여 RT-PCR을 수행하였다. 사용한 억제제로서, Antipain (sigma Aldrich), chymostatin (sigma Aldrich), E-64 (sigma Aldrich), E-64 (Santa Cruz Biotechnology, Inc.), epoxomicin (sigma Aldrich), MG132(sigma Aldrich)을 사용하였고, 모두 DMSO에 녹였다. Ⅲ. 결과 A. TCM-33 추출물이 바이러스 생산에 미치는 영향 TCM-33 추출물이 MHV-A59에 항바이러스 효과가 있는지 알아보았다. TCM-33 추출물을 처리했을 때, 바이러스 생산에 어떠한 영향을 미치는지 알아보기 위하여, DBT 세포에 2MOI의 MHV-A59를 감염시킬 때 TCM-33 추출물을 처리하였다. 12시간 동안 배양 후에, 바이러스가 방출된 배양액을 수확한 뒤 plaque assay를 하여 바이러스의 titer를 관찰하였다. TCM-9 추출물은 MHV-A59에 억제효과가 없다고 밝혀진 약재로서 음성 대조군으로 사용하였고, ribavirin은 항바이러스제로 널리 알려진 약재로서 양성 대조군으로 사용하였다. 바이러스의 titer가 TCM-33 추출물의 농도에 따라 감소하였다. TCM-33 추출물을 50 ㎍/ml로 처리했을 때, 바이러스만 처리했을 때와 비교하여 3 log 이상 감소를 하였고, 100 ㎍/ml로 처리했을 때는 5 log 이상 감소하였다. 또한 TCM-33 추출물은 항바이러스 효과가 있다고 보고된 ribavirin보다 50 ㎍/ml로 처리했을 때는 1 log 이상, 100 ㎍/ml로 처리했을 때는 2 log 이상 더 감소하였다 (Fig. 3).
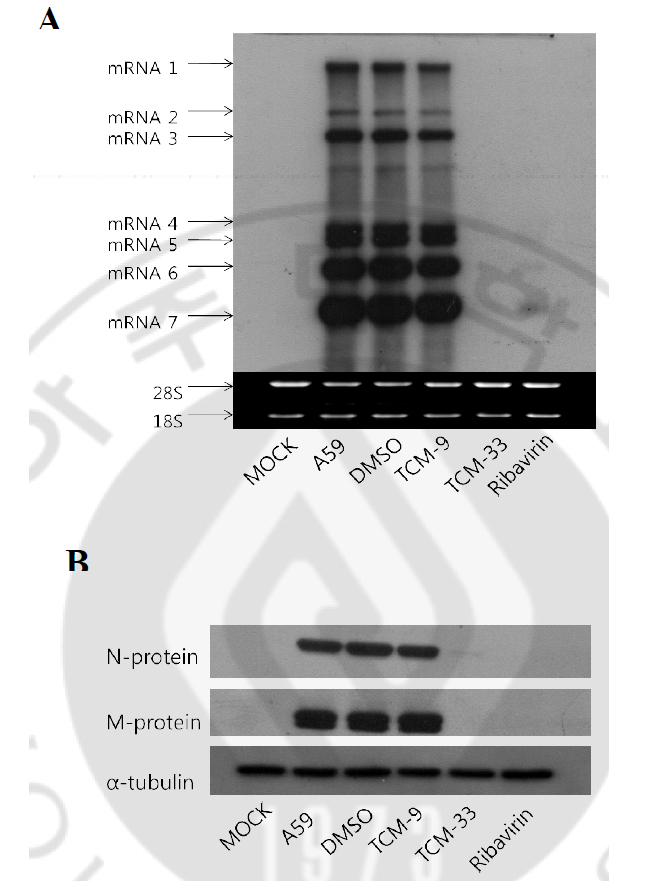
Fig. 3. Does-dependent inhibition of MHV-A59 replication by TCM-33 extract. Varying concentrations (1,10,30,50,70,100㎍/ml) of TCM-33 extract was applied to DBT cells, while they were simultaneously infected with MHV-A59 at 2MOI, for 12hr at 37 ℃. The virus titer was measured by plaque assay. Error bars represent the standard deviations from three independent experiments. 바이러스 생산이 TCM-33 추출물에 의하여 현저히 감소하였다. 그래서 TCM- 33 추출물이 바이러스의 mRNA나 단백질 합성에도 영향을 미치는지 알아보았다. 세포에 바이러스를 감염시킬 때, 동시에 TCM-33 추출물을 50㎍/ml으로 처리한 뒤, 5시간 30분 동안 배양하였다. 바이러스의 mRNA와 단백질 합성을 Northern blot과 Western blot으로 관찰하였다. Northern blot에 사용된 probe는 바이러스 gene 7에 특이적으로 결합하는 probe로서 mRNA1에서부터 mRNA7까지 관찰할 수 있다. 바이러스가 감염되었을 때, 바이러스의 mRNA 1에서 mRNA 7까지 관찰되었으나, TCM-33 추출물을 처리하였을 때, 바이러스의 mRNA 수준이 현저히 감소하였고, ribavirin보다 더 감소 효과를 보였다 (Fig. 4A). 바이러스의 mRNA 수준에서 관찰한 결과와 동일한 결과를 바이러스의 단백질 합성에서도 관찰되었다. TCM-33 추출을 처리하였을 때, 바이러스의 N 단백질과 M 단백질이 현저히 감소하였다 (Fig. 4B). 따라서 TCM-33 추출물이 바이러스의 mRNA의 합성을 억제하여 단백질 합성이 억제되었고, 최종적으로 바이러스의 생산이 감소되었다고 추정하였다.
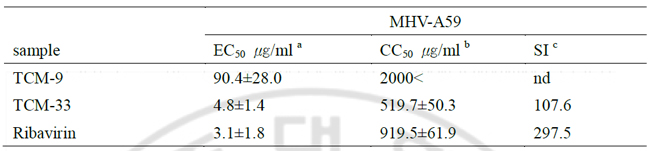
Fig.4. Effect of TCM-33 extract on MHV-A59 RNA and protein expression. (A) Northern blot to detect MHV-A59 RNA expression in response to treatment with 50 ㎍/ml of TCM-33 extract. Intracellular RNAs from mock-infected, MHV- infected without TCM-33 extract, MHV-infected with DMSO, MHV- infected with ribavirin, MHV- infected with TCM-9 extract, MHV- infected with TCM-33 extract were extracted at 5hr 30min postinfection and analyzed by northern blot analysis using a gene-7-specific probe. Seven species of mRNAs and 28S and 18S ribosomal RNAs are indicated. (B) Western blot analysis to detect MHV-A59 nucleocapsid (N) protein and membrane (M) protein in response to treatment with 50 ㎍/ml of TCM-33 extract. Lysates at 5hr 30min post-infection were subjected to 10% SDS-PAGE, transferred to PVDF membranes and incubated with monoclonal antibodies against NHV N and M protein. TCM-33 추출물의 효과를 알아보기 위하여, 50% effective concentration (EC50)와 50% cytotoxic concentration (CC50)를 계산하였다. EC50는 약재에 의하여 바이러스 titer 가 50% 감소되는 농도를 말하는 것으로, TCM-9 추출물은 90.4±28.0 ㎍/ml, TCM-33 추출물은 4.8±1.4 ㎍/ml, ribavirin 은 3.1±1.8 ㎍/ml이다 (Table 2). CC50는 약재에 의하여 세포의 생존능력 (viability)이 50% 감소되는 농도를 말하는 것으로, TCM-33 추출물은 519.7±50.3 ㎍/ml, ribavirin 은 919.5±61.9 ㎍/ml 이였으나, TCM-9 추출물의 CC50 값은 농도를 2000 ㎍/ml까지 처리하였는데도 불구하고 구하지 못하였다. selectivity index (SI: CC50/EC50)는 바이러스에 대한 약재의 항바이러스 효과를 나타내며, TMC-33 추출물은 107.6이며, ribavirin의 경우 297.5이다. 일반적으로 약재에 의하여 바이러스 생산이 10%로 감소되면 항바이러스 효과가 있다고 말한다. 그래서 약재의 EC90 값을 알아보니 TCM-33 추출물은 17.3±2.3 ㎍/ml이고, Ribavirin 은 26±5.6 ㎍/ml이다. 세포의 생존능력 (viability)은 MTT assay 이외에, 약재에 의하여 세포의 증식 (proliferation)에 변화가 있는지를 세포의 개체 수가 두 배로 늘어나는 시간을 비교하여 관찰하였다. TCM-33 추출물을 처리하였을 때, 세포의 증식에 크게 영향을 보이지 않았다. 따라서 TCM-33 추출물이 MHV-A59에 대하여 효과적인 약재라고 추정되며, 항코로나바이러스 약재의 가능성을 제시하였다. Table 2. Effect of TCM-33 extract on MHV-A59
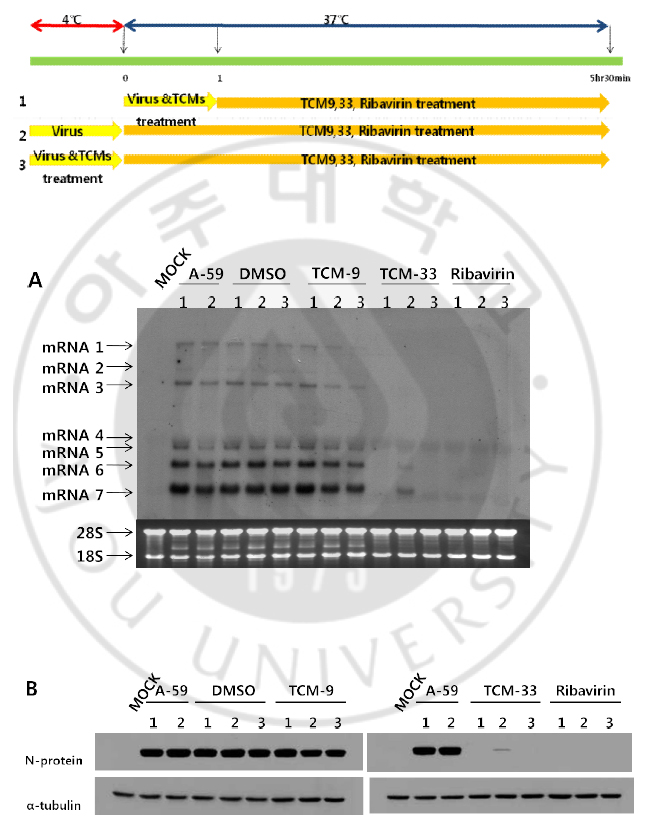
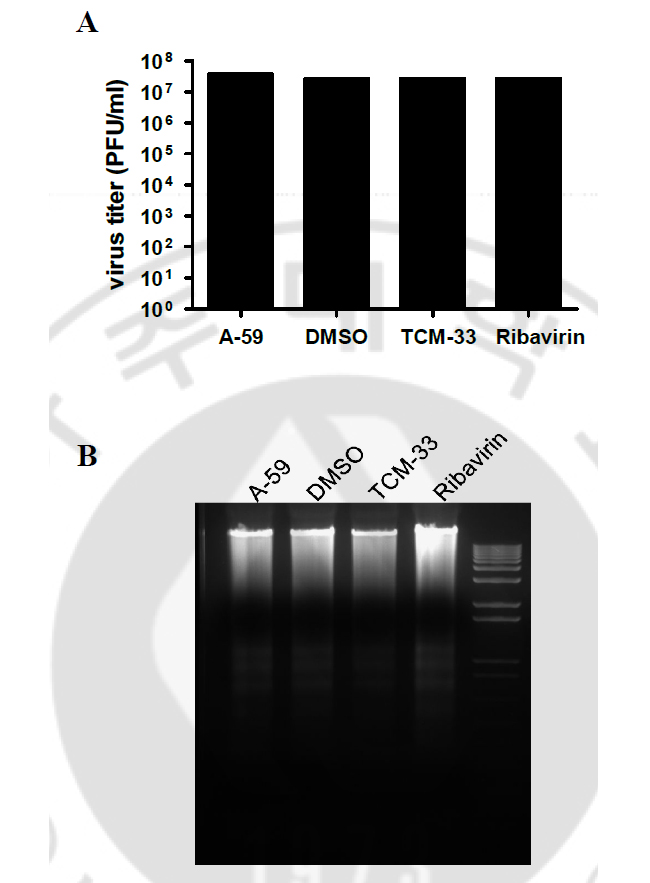
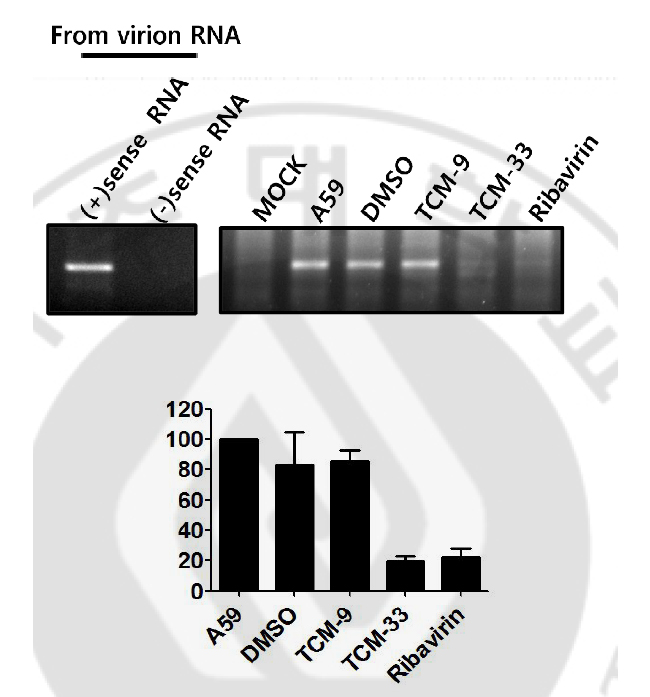
each value represents the mean ±SD from three independent experiments. aThe 50% effective concentration (EC50) was determined as the concentration of extract needed to inhibit the virus titer by 50% of the control value (cell without addition of extract). bThe 50% cytotoxic concentrarion (CC50) was determined as the concentration of extract necessary to reduce the cell viability to 50% of the control (cell without addition of extract). cThe selectivity index (SI) was calculated by CC50/EC50. MHV-A59, mouse hepatitis virus A59 strain. nd, not detected B. TCM- 33 추출물이 바이러스 진입 (entry)에 미치는 영향 앞에서, TCM-33 추출물이 바이러스의 mRNA 합성을 억제하였으므로, TCM-33 추출물은 바이러스 증식에서 mRNA의 합성 전 단계에서 영향을 미칠 것이라 생각하였다. MHV-A59은 숙주세포 막에 발현되어 있는 CEACAM-1 수용체와 virion에 발현되어 있는 S 단백질과의 상호작용에 의하여 바이러스가 숙주세포 내로 들어오게 되고, 바이러스는 자신의 유전체를 세포질에 방출한다. 그래서 TCM-33 추출물이 바이러스의 진입에 영향을 주는지 알아보았다. TCM-33 추출물이 바이러스 진입에 영향을 미치는지 알아보기 위하여, DBT 세포에 MHV-A59을 4℃에서 1시간 동안 처리하여 바이러스가 숙주세포 막에만 부착될 수 있게 하였고, 부착되지 않은 바이러스는 세척하여 제거하였다. 그 후에 TCM-33 추출물을 50㎍/ml로 처리하여 37℃에서 배양하여 숙주세포 막에 부착되어 있던 바이러스가 동시에 세포 내로 들어갈 수 있게 하였다. 다른 대조군으로 DBT 세포에 MHV-A59을 4℃에서 1시간 동안 처리할 때, 동시에 TCM-33 추출물을 처리한 후에 37℃에서 배양하여, 직접적으로 TCM-33 추출물에 의한 바이러스 진입에서의 영향을 알아보았다. TCM-33 추출물이 바이러스 진입에 영향이 있다면, TCM-33 추출물을 처리했을 때보다 4℃에서 바이러스만을 처리했을 때, 바이러스의 mRNA와 단백질 합성이 더 높을 것이라고 예상하였다. 37℃에서 바이러스를 처리했을 때 보다 4℃에서 처리했을 때, 바이러스의 mRNA의 발현 수준이 약간 낮게 관찰되었다. DMSO, TCM-9 추출물과 ribavirin을 처리했을 때, 각각의 대조군에서 바이러스의 mRNA의 발현 수준의 변화는 없었다. 그러나 TCM-33 추출물을 처리했을 때, 4℃에서 바이러스만 처리한 세포에서 바이러스의 mRNA 발현 수준이 더 강하였다 (Fig. 5A). 바이러스의 mRNA에서와 마찬가지로, 바이러스의 단백질 합성도 동일한 결과를 보였다 (Fig. 5B). 따라서 TCM-33 추출물이 바이러스 진입에 부분적으로 영향을 미칠 것이라 추정하였다. 그러나 바이러스의 진입을 완전히 확인한 것이 아니라 바이러스가 세포에 부착하는 단계를 본 것이고, TCM33 추출물은 약하지만 세포독성이 있으므로 약재가 바이러스의 감염력에 미치는 영향을 배제할 수 없다. 따라서 TCM-33 추출물이 바이러스에 직접적인 영향을 미치는지 알아보기 위하여 바이러스에 약재를 먼저 처리한 후에 세포에 감염시켜서 바이러스의 titer를 알아보았다. 약재를 처리하지 않았을 때와 약재를 처리하였을 때를 비교하면, 바이러스 titer의 변화가 없었다 (Fig. 6A). 또한 바이러스의 RNA에 약재가 직접적으로 영향을 주는지 알아보기 위하여 virion으로부터 바이러스의 RNA를 추출하여, RNA에 약재를 처리하여 약재에 의한 바이러스의 RNA의 영향을 알아보았다. 약재에 의하여 바이러스의 RNA가 영향을 받은 것은 아니라는 것을 관찰할 수 있었다 (Fig. 6B). 이 결과로 약재는 직접적으로 바이러스의 감염력 또는 바이러스의 RNA에 영향을 주지 않을 것이라 추정하였다.
Fig. 5. Inhibitory effects of the TCM-33 extract on MHV-A59 entry. (A) Northern blot to detect MHV-A59 RNA expression in response to treatment with 50 ㎍/ml of TCM-33 extract. Intracellular RNAs from mock-infected, MHV- infected without TCM-33 extract, MHVinfected with DMSO, MHV- infected with ribavirin, MHV- infected with TCM-9 extract, MHV- infected with TCM-33 extract that MHV treatment at 4℃ and temperature shift 37℃, or 37℃ were extracted at 5hr 30min post-infection and analyzed by northern blot analysis using a gene-7-specific probe. Seven species of mRNAs and 28S and 18S ribosomal RNAs are indicated. (B) Western blot analysis to detect MHV-A59 nucleocapsid (N) protein and membrane (M) protein in response to treatment with 50 ㎍/ml of TCM-33 extract. Lysates at 5hr 30min post-infection were subjected to 10% SDS-PAGE, transferred to PVDF membranes and incubated with monoclonal antibodies against NHV N and M protein.
Fig. 6. Direct effect of TCM-33 extract on MHV-A59 entry. (A) To direct effect of TCM-33 extract on MHV-A59 entry, virus were pretreatment with TCM-33 extract for 1hr at RT then 10 fold diluted virus infected to DBT cell for 12hr at 37℃. The virus titer was measured by plaque assay. (B) To degradation of virion RNA by TCM-33 extract, genomic RNA from partially purified viron incubated with TCM-33 extract for 1hr at RT. C. TCM-33 추출물에 의한 음성가닥 RNA 합성의 영향 TCM-33 추출물이 MHV-A59 증식 단계 중 어느 단계에서 주요하게 영향을 미치는지 알아보았다. MHV-A59의 증식에서 mRNA의 합성 전 단계가 음성가닥 RNA의 합성이다. 바이러스는 유전체로부터 음성가닥 RNA를 합성한 뒤, 양성가닥 RNA와 subgenomic mRNA를 합성한다. 그래서 TCM-33 추출물이 음성가닥 RNA 합성에 영향을 미치는지 알아보았다. 음성가닥 RNA의 합성은 바이러스가 세포 내로 들어가서 증식이 일어나는 과정에서 볼 때, 세포 내로 들어와서 빠른 시간 안에 일어날 것이라 생각하였다. 그래서 DBT 세포에 2MOI의 MHV-A59을 4℃에서 1시간 동안 처리하여 TCM-33 추출물에 의한 바이러스 진입 과정에서의 억제효과를 배제하였다. 그런 다음 37℃에서 배양한 후, 전체 RNA을 추출하여 RT-PCR을 통하여 음성가닥 RNA 합성을 알아보았다. RT-PCR을 통해서 음성가닥 RNA를 관찰하는데, 음성가닥 RNA만을 선택적으로 관찰할 수 있게 하기 위하여 rimer를 제작하였다. 그런데 이 primer가 선택적으로 음성가닥 RNA만을 관찰할 수 있는지 알아보기 위하여, virion으로부터 추출한 바이러스의 genomic RNA를 대조군으로 사용하였다. virion으로부터 바이러스의 genomic RNA의 추출은 sucrose gradient를 통해서 virion을 분리한 후, virion으로부터 genomic RNA를 추출하였다. Virion으로부터 추출한 바이러스의 genomic RNA는 양성가닥 RNA이기 때문에 음성가닥 RNA는 확인할 수 없다. 바이러스가 세포 내로 들어오고 나서 어느 시간에서 음성가닥 RNA가 관찰되는지를 알아보기 위하여 1시간, 2시간, 3시간 동안 배양하였다. 바이러스가 세포 내로 들어온 후, 3시간 배양하였을 때 가장 현저하게 음성가닥 RNA를 관찰할 수 있었다. 따라서 TCM-33에 의한 음성가닥 RNA 합성의 영향을 보기 위한 실험에서는 3시간 동안 배양하였다. TCM-33 추출물에 의한 음성가닥 RNA 합성의 영향을 알아보기 위하여, 바이러스를 배양하는 동안 약재를 같이 처리하였다. virion으로부터 추출한 바이러스의 genomic RNA는 양성가닥 RNA만이 확인되고, 음성가닥 RNA는 관찰되지 않는 것을 확인하였다. 바이러스가 감염된 세포에서는 음성가닥 RNA의 합성이 강하게 이루지는 것을 볼 수 있는데, TCM-33 추출물을 처리했을 때는 음성가닥 RNA의 합성이 현저히 감소하였다 (Fig. 7). 바이러스만 감염되었을 때와 비교하면, TCM-33을 처리하였을 때 대략 80% 정도 감소하였다. 결과적으로 음성가닥 RNA의 합성이 TCM-33 추출물에 의하여 현저히 감소하였기에 TCM-33 추출물이 음성가닥이 합성되기 전 단계에서 영향을 미칠 것이라 추정하였다.
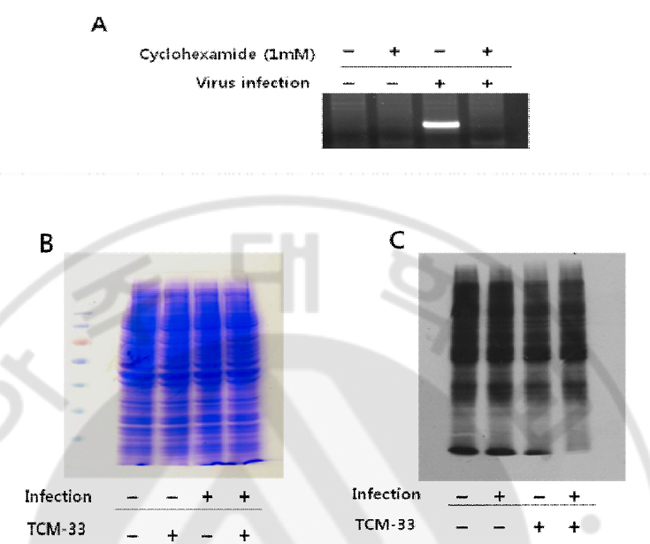
Fig. 7. Inhibitory effect of the TCM-33 extract on the minus-strand RNA synthesis. Reverse transcriptase (RT)-PCR was used to detect viral minus-strand RNA expression. MHV-A59 treatment to DBT cell of 2MOI for 1hr at 4℃. Temperature shift at 37℃ 50㎍/ml of TCM-33 extract. Total RNA extracted at 3hr post-infection and analyzed by RT-PCR using minus-strand RNA specific probe. The mean ±SD of RNA expression levels from five independent experiments are shown. D. TCM-33 추출물에 의한 세포의 단백질 합성의 영향 TCM-33 추출물이 바이러스의 음성가닥 RNA 합성을 억제시켰다. 따라서 TCM-33 추출물이 음성가닥 RNA 합성 이전 단계에서 영향을 미칠 것이라 추정하였다. MHV-A59는 숙주세포 내로 들어오면 바로 번역이 일어난다. 이때의 번역은 숙주세포의 translation machinery를 이용하여 일어난다. 그래서 세포의 단백질 합성을 억제하면 초기 바이러스의 번역도 억제가 되는지를 알아보았다. 번역 억제제로서 잘 알려진 cyclohexamide를 처리하여 세포의 번역을 억제하였다. Cyclohexamide를 처리하였을 때, 바이러스의 음성가닥 RNA의 합성이 이루어지지 않는 것을 관찰하였다 (Fig. 8A). 따라서 세포의 번역을 억제하면 바이러스의 증식 또한 억제되며, 초기 바이러스의 번역은 세포의 translation machinery를 이용한다는 것을 확인할 수 있었다. TCM-33 추출물이 초기 바이러스의 번역에 영향을 미치는지 알아보았다. 그러나 직접적으로 초기 바이러스의 번역을 보는 것은 불가능하여, 간접적으로 숙주세포의 단백질 합성의 영향을 알아보았다. 세포의 단백질 합성의 영향을 알아보기 위하여, DBT 세포에 2MOI의 MHV-A59을 감염시킬 때, TCM-33 추출물을 처리를 하였다. 3시간 배양 후에, lysis buffer를 이용하여 세포를 용해시켰다. 용해물을 10% SDS-PAGE 한 후, gel을 coomassie blue을 사용하여 세포의 총 단백질 합성을 관찰하였다. 바이러스가 감염되지 않은 세포와 바이러스가 감염된 세포 간의 단백질 합성의 차이는 보이지 않았고, 또한 TCM-33 추출물을 처리하였을 때도 바이러스 감염 여부와 상관없이 세포의 단백질 합성에는 영향이 없었다 (Fig. 8B). Coomassie blue를 이용하여 본 세포의 단백질은 세포의 총 단백질을 본 것이기 때문에, 바이러스가 감염되고 나서 새로이 합성되는 단백질의 영향을 알아보았다. DBT 세포에 2MOI의 HV-A59을 4℃에서 1시간 동안 처리한 후, TCM-33 추출물을 처리하여 37℃에서 2시간 10분 동안 배양하였다. 바이러스가 감염된 후에 20분간의 세포의 단백질 합성의 영향을 알아보기 위하여 methionine free media를 37℃에서 30분간 처리한 후에, 100μci/ml의 L-35S methionine을 37℃에서 20분간 pulse labeling 한 뒤, 10% SDSPAGE 하여 단백질 합성을 관찰하였다. 바이러스가 감염되었을 때와 바이러스가 감염되지 않았을 때, 30분간의 새로이 합성된 단백질의 합성은 차이가 없었다. 또한 TCM-33 추출물을 처리하였을 때도, 바이러스 감염 여부에 상관없이 새로이 합성된 단백질의 합성에는 차이가 없었다 (Fig. 8C). 따라서 TCM-33 추출물은 세포의 단백질 합성에는 영향을 주지 않는다고 생각되며, 또한 초기 바이러스의 번역에도 영향을 미치지 않을 것이라고 추정하였다. 결과를 종합해보면, TCM-33 추출물은 바이러스가 세포질에서 초기에 일어나는 번역에는 영향을 미치지 않을 것이라 생각되며, 결국에는 번역된 뒤에 일어나는 과정에서부터 음성가닥 RNA가 합성되는 과정에 영향을 미칠 것이라 추정하였다.
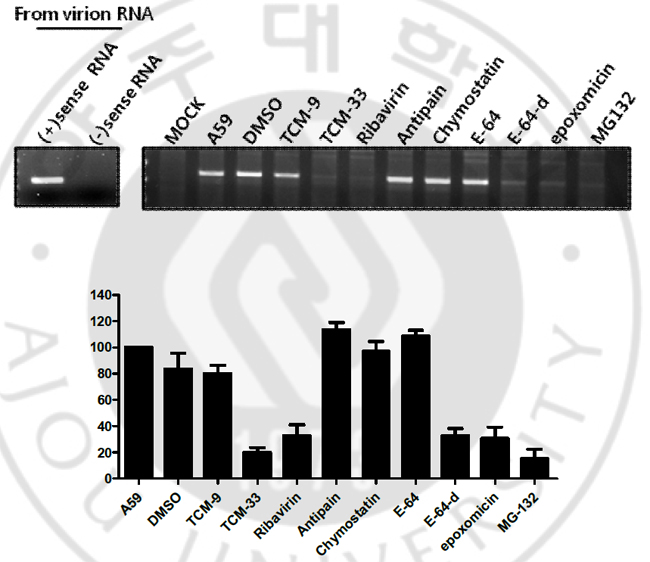
Fig. 8. Affect of intracellular protein translation by TCM-33 extract treatment. (A) Reverse transcriptase (RT)-PCR was used to detect viral minus-strand RNA expression. DBT cell infected 2MOI of MHV-A59 at 4 for 1hr and then temperature shift ℃ at 37℃ with 1mM cyclohexamide. Total RNA extracted at 3hr post-infection and analyzed by RT-PCR using minus-strand RNA specific probe. (B) Coomassie blue staining to detect cellular protein translation in the TCM-33 extract. DBT cell infected 2MOI of MHV-A59 at 4℃ for 1hr and then temperature shift at 37℃ with TCM-33 extract. Lysates at 3hr post-infection were subjected to 10% SDS-PAGE, coomassie blue stain. (C) metabolic labeling to detect cellular protein translation for 30min in the TCM-33 extract. DBT cell infected 2MOI of MHV-A59 at 4℃ for 1hr and then temperature shift at 37℃ with TCM-33 extract for 2hr 10min and then change the methionine free media at 37℃ for 30min. L-35S methionine (100μci/ml) treatment at 37℃ for 20min. lyastes were subjected to 10% SDS-PAGE, destainnig solution at 25℃for 1hr, enhance solution 25℃ for 1hr, drying at 80℃ for 90min. E. non-structural protein processing에 의한 RNA 합성 MHV-A59의 증식에서 음성가닥 RNA의 전 단계는 번역되어 합성된 polypeptide의 non-structural protein processing이다. non-structural Protein processing은 바이러스가 자신의 유전체에 암호하고 있는 proteinase에 의하여 일어난다. MHVA59은 유전체에 3가지의 proteinase을 가지고 있다 (PLP1, PLP2, 3CLpro). PLP1과 PLP2는 papain-like proteinase이고, 3CLpro는 chymotrypsin-like proteinase이며, PLP1과 PLP2는 polypeptide의 nsp1~4 부분을 자르고, 3CLpro는 polypeptide의 나머지 부분을 자른다. non-structural Protein processing이 일어나지 않게 되면, 음성가닥 RNA 합성이 일어나지 않는지 알아보았다. 이를 위해 cystein/serine proteinase의 억제제로 알려진 antipain, chymostatin, E-64, E64-d를 사용하였고, proteasome의 chymotrypsin-like prteinase의 억제제로 알려진 epoxomicin과 MG132를 사용하였다. 각각의 억제제를 처리하였을 때, 음성가닥 RNA의 합성이 억제되는지를 알아보기 위하여, DBT 세포에 MHV-A59을 4℃에서 1시간 동안 처리한 뒤, 억제제를 37℃에서 3시간 동안 처리하여 음성가닥 RNA의 합성을 관찰하였다. Antipain, chymostatin, E-64를 처리하였을 때, 바이러스의 음성가닥 RNA의 합성은 감소하지 않았지만, E-63-d, epoxomicin, MG132를 처리하였을 때에는 바이러스의 음성가닥 RNA의 합성이 현저히 감소하였다. 바이러스만을 감염시켰을 때와 비교하여, E-64-d와 epoxomicin은 70% 감소하였고, MG132는 80% 정도 감소하였다 (Fig. 9). E-64와 E-64-d는 동일한 억제제이나 E-64-d가 세포 투과성이 더 높은 억제제이다. 그래서 E-64는 음성가닥 RNA가 감소하지 않았지만, E-64-d는 세포에 대한 투과성이 높아 음성가닥 RNA의 합성에 보다 효과적으로 억제효과를 보이는 것으로 생각되었다. 이 결과로 proteinase의 활성이 억제되면, 음성가닥 RNA의 합성이 현저하게 감소하는 것을 관찰하였다. 따라서 음성가닥 RNA의 합성은 protein processing이 먼저 일어난 후 합성된다는 것을 확인하였다.
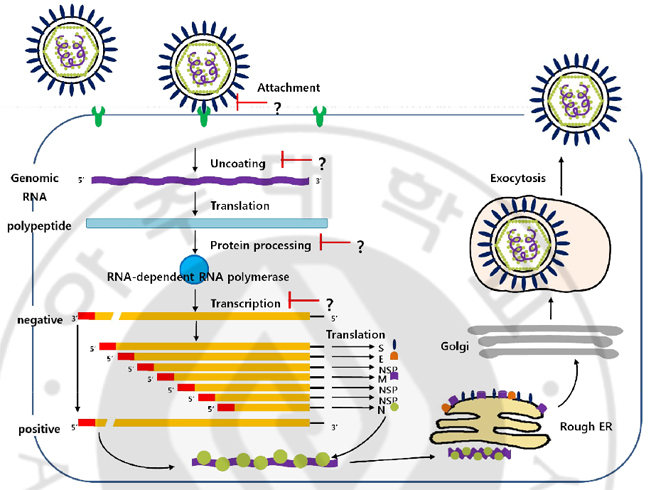
Fig. 9. Minus-strand RNA synthesis by non-structural protein processing. Reverse transcriptase (RT)-PCR was used to detect viral minus-strand RNA expression in the proteinase inhibitors. MHV-A59 treatment to DBT cell of 2MOI for 1hr at 4℃. Temperature shift at 37℃ 50㎍/ml of TCM-33 extract, Ribavirin and proteinase inhibitors. Total RNA extracted at 3hr post-infection and analyzed by RT-PCR using minus-strand RNA specific probe. The mean ±SD of RNA expression levels from five independent experiments are shown. Ⅳ. 고찰 SARS가 창궐한 후에 코로나바이러스 감염에 대한 치료 연구가 활발해졌다 (Haagmans and Osterhaus, 2006; Stockman et al., 2006). 그중, traditional Chinese medicine (TCM)으로부터 얻은 몇몇 성분들과 여러 개의 작은 허브 성분이 in vitro에서 항 SARS 코로나바이러스 효과가 있다고 밝혀졌고, 이 논문에서 TCM이 SARS 환자에서도 효과가 있다고 보고 하였다(Cinatl et al., 2003; Chen et al., 2004; Wu et al., 2004; Cinatl et al., 2005). 최근에 승마 (cimicifuga fhizoma), 고련피 (meliae cortex), 황련 (coptidis rhizoma), 황백 (phellodendeon cortex), 고삼 (sophorae radix), 오가피 (acanthopanacis cortex), 지유 (sanguisorbae radix), 사상자 (torilis fructus)가 항코로나바이러스 효과가 있다고 보고하였다(Kim et al., 2008; Kim et al., 2010). 그러나 아직까지 이런 항코로나바이러스 효과가 있는 약재들이 어떠한 기작을 통해 효과를 보이는지에 대한 것은 밝혀지지 않았다. 그래서 새로운 항코로나바이러스 약재를 찾아내기 위하여 TCM-33 추출물을 이용하여 항코로나바이러스 효과와 그 기작을 알아보았다. 본 연구에서 TCM-33 추출물이 코로나바이러스 중 MHV-A59 바이러스의 증식을 효과적으로 감소시키는 것을 확인하였고 (Table 1), 이러한 감소 효과는 바이러스의 mRNA와 단백질 합성이 감소되어 나타난 것이라는 것을 밝혔다 (Fig 4). TCM-33 추출물이 MHV-A59에 보인 항바이러스 효과가 바이러스 증식에서 보았을 때, 어느 단계에서 억제효과를 보이는지 알아보았다. Fig 5.에서 TCM-33 추출물이 바이러스가 세포막에 부착하는 단계에서 약하지만 영향을 미치는 것을 관찰하였다. 그러나 이러한 TCM-33의 억제효과는 Fig 4에서 확인하였던 억제효과를 생각하면 충분한 억제효과를 보인다고는 볼 수 없고, TCM-33이 세포에 약간의 세포독성을 가지고 있고, 바이러스의 감염력 또는 바이러스의 RNA에 영향을 미치지 않을 것이라 추정하였다. 따라서 바이러스가 세포막을 통과하여 세포질로 들어가고 나서의 과정에서 TCM-33이 영향을 미칠 것이라 생각하였다. 바이러스가 세포로 들어와서 바로 일어나는 과정은 번역으로, 번역은 숙주세포의 translation machinery를 이용하여 일어난다. 그러나 TCM-33은 세포의 단백질 합성에도 영향을 주지 않았고, 바이러스가 감염되고 나서 새로이 합성된 단백질에도 영향을 주지 않았다 (Fig. 8). 따라서 TCM-33 추출물이 초기 바이러스의 번역에 영향을 미치지 않을 것이라 추정하였다. TCM-33이 초기 바이러스의 번역에 영향을 주지 않는다면, non-structural protein processing이나 RdRp에 의한 음성가닥 RNA 합성에 영향을 미칠 것이라 추측하였다. Fig. 7에서 TCM-33 추출물에 의하여 바이러스의 음성가닥 RNA 합성이 현저히 감소하는 것을 관찰하였다. 결과적으로 TCM-33은 바이러스가 세포질에서 번역이 일어난 다음 단계: non-structural protein processing, RdRp의 활성에서 영향을 미칠 것이라 추정하였다 (Fig 10.).
Fig. 10. Proposed model for inhibition of MHV-A59 replication by TCM-33 extract. TCM-33 extract might inhibit early stage of MHV-A59 replication, which involved entry, translation, protein processing, negative-strand RNA synthesis. 최근 아연이온 (Zn2+)이 코로나바이러스와 arterivirus의 RNA polymerase 활성을 억제시키고, zinc ionophores가 이런 바이러스의 증식을 방해한다고 보고되었다. 이 논문에서 아연이온이 eqine arteritis virus (EAV)의 RNA 합성의 개시 단계에 선택적인 억제제라는 것을 보여주었고, SARS 코로나바이러스의 경우에는 RNA 합성의 신장 단계를 억제하여 증식을 억제한다고 보고하였다. 또한 SARS 코로나바이러스에서의 아연의 효과가 RdRp가 주형에 결합하는 단계에 직접적으로 영향을 주어 나타나는 것이라고 말하고 있으며, SARS 코로나바이러스의 RdRp의 3차원적 구조에 아연이 결합할 수 있는 특정 부분 (motif)이 있다는 것을 밝혔다. 따라서 아연이 RdRp에 결합하여 RdRp가 주형과의 결합을 방해하는지에 대한 것을 추후 더 연구를 해야 할 것이라고 보고를 하였다 (te Velthuis et al., 2010). 본 연구에 사용된 TCM-33 추출물 또한 이 논문에서처럼 MHV-A59의 RNA 합성 과정에서 RdRp의 활성에 영향을 주는지에 대한 것은 추후에 더 실험을 해야 할 것이다. 코로나바이러스의 RNA 합성은 polypeptide가 proteinase에 의하여 protein processing이 일어난 후, RNA 합성이 일어난다. 그래서 Fig 6에서 본 음성가닥 RNA 합성의 감소는 RdRp의 활성에 의한 것 일 수도 있지만 protein processing이 일어나지 않아서 나타난 결과일 수 있다고 추측하였다. protein processing은 바이러스가 유전체에 암호되어 있는 proteinase에 의하여 일어나며, 현재까지 알려진 바로는 PLP1/2에 의하여 nsp1~3의 단백질이 형성되며 (Schiller et al., 1998; Brockway et al., 2004; Harcourt et al., 2004; Brockway and Denison, 2005), 3CL에 의하여 나머지 nsp가 형성된다 (Denison et al., 1998). RNA 합성에 필요한 RdRp는 nsp12에 암호화되어 있기 때문에 3CL에 의하여 protein processing이 일어나야 한다. 하지만 효율적인 RNA 합성이 이루어지기 위해서는 3CL에 의한 protein processing만이 일어나는 것이 아니라 PLP1/2에 의한 protein procesisng도 같이 일어나야 효율적인 RNA 합성이 이루어질 수 있다 (Gadlage et al., 2010). 이전에 cysteine proteinase inhibitor인 E64d가 코로나바이러스의 protein processing과 RNA 합성을 억제한다는 보고 있었다 (Kim et al., 1995). 본 실험에서 사용된 TCM-33 추출물이 이런 protein processing에 관여하는지에 대한 내용은 추후에 더 실험을 해야 할 것이다. Proteasome 억제제로 사용되었던 MG132가 MHV-JHM의 entry를 억제한다고 보도된 바가 있다 (Yu and Lai, 2005). MG132가 세포 내에 존재하면, MHV-JHM이 세포 내로 들어와서 세포질로 방출되지 않아 증식을 할 수 없게 됨을 밝혔으며, MG132에 의하여 바이러스가 세포 내로 들어오는 과정 중의 세포 내 이입 (endocytosis)을 통하여 들어오며 엔도좀 (endosome)과 리소좀 (lysosome)에 머무르게 하고 세포질로의 방출을 억제하는 것을 밝혔다. 따라서 unbiqutinproteasome system이 바이러스의 진입에 관여할 것이라고 제시하였으며, 이러한 영향은 바이러스 중에서도 세포 내 이입을 이용하는 바이러스의 한에서 이루어질 것이라고 제시하였다. MHV-A59은 세포 내 이입을 이용하지 않기 때문에 이러한 영향을 관찰할 수 없었다고 하였다. 따라서 Fig. 9.에서 proteasome 억제제를 처리하였을 때, 바이러스의 음성가닥 RNA 합성의 감소는 억제제가 바이러스의 진입에 영향을 미쳐서 나타난 결과가 아닐 것이라고 추정하였지만 정확하게 알아보는 실험을 추후에 해야 할 것이다. 종합하면, TCM-33 추출물이 MHV-A59의 증식을 효과적으로 억제하였고, TCM-33 추출물이 바이러스가 세포질에서 초기 번역이 된 후, 형성되는 polypeptide의 protein processing과/또는 RdRp의 활성에 영향을 미쳐서 음성가닥 RNA 합성을 감소시킨 것이라 추정하였다. Ⅴ. 결론 본 연구에서, TCM-33 추출물이 MHV-A59의 증식을 효과적으로 억제하였다. TCM-33 추출물은 MHV-A59의 증식 과정 중에서 바이러스 진입 (부착 단계)에 부분적으로 효과를 보일 것이라 추정하였다. 또한 TCM-33 추출물은 음성가닥 RNA 합성을 현저히 억제하였으며, proteinase 억제제를 통하여 protein processing 후에 음성가닥 RNA 가 합성됨을 보았다. 그러나 TCM-33 추출물은 세포의 단백질 합성에는 아무런 영향이 없었다. 따라서 TCM-33 추출물은 MHVA59 증식의 초기 단계 (early stage)에 영향을 미칠 것이라 생각되며, 아마도 protein processing 과/또는 RdRp의 활성일 것이라고 추정하였다. 참고문헌 1. Balzarini J, Keyaerts E, Vijgen L, Vandermeer F, Stevens M, De Clercq E, Egberink H, Van Ranst M: Pyridine N-oxide derivatives are inhibitory to the human SARS and feline infectious peritonitis coronavirus in cell culture. J Antimicrob Chemother 57: 472-481, 2006. 2. Bredenbeek PJ, Pachuk CJ, Noten AF, Charite J, Luytjes W, Weiss SR, Spaan WJ: The primary structure and expression of the second open reading frame of the polymerase gene of the coronavirus MHV-A59; a highly conserved polymerase is expressed by an efficient ribosomal frameshifting mechanism. Nucleic Acids Res 18: 1825-1832, 1990. 3. Brockway SM, Denison MR: Mutagenesis of the murine hepatitis virus nsp1-coding region identifies residues important for protein processing, viral RNA synthesis, and viral replication. Virology 340: 209-223, 2005. 4. Brockway SM, Lu XT, Peters TR, Dermody TS, Denison MR: Intracellular localization and protein interactions of the gene 1 protein p28 during mouse hepatitis virus replication. J Virol 78: 11551-11562, 2004. 5. Chen F, Chan KH, Jiang Y, Kao RY, Lu HT, Fan KW, Cheng VC, Tsui WH, Hung IF, Lee TS, Guan Y, Peiris JS, Yuen KY: In vitro susceptibility of 10 clinical isolates of SARS coronavirus to selected antiviral compounds. J Clin Virol 31: 69-75, 2004. 6. Chen Y, Cai H, Pan J, Xiang N, Tien P, Ahola T, Guo D: Functional screen reveals SARS coronavirus nonstructural protein nsp14 as a novel cap N7 methyltransferase. Proc Natl Acad Sci U S A 106: 3484-3489, 2009. 7. Cinatl J, Jr., Michaelis M, Hoever G, Preiser W, Doerr HW: Development of antiviral therapy for severe acute respiratory syndrome. Antiviral Res 66: 81-97, 2005. 8. Cinatl J, Morgenstern B, Bauer G, Chandra P, Rabenau H, Doerr HW: Glycyrrhizin, an active component of liquorice roots, and replication of SARS-associated coronavirus. Lancet 361: 2045-2046, 2003. 9. Decroly E, Imbert I, Coutard B, Bouvet M, Selisko B, Alvarez K, Gorbalenya AE, Snijder EJ, Canard B: Coronavirus nonstructural protein 16 is a cap-0 binding enzyme possessing (nucleoside-2'O)-methyltransferase activity. J Virol 82: 8071- 8084, 2008. 10. Deming DJ, Graham RL, Denison MR, Baric RS: Processing of open reading frame 1a replicase proteins nsp7 to nsp10 in murine hepatitis virus strain A59 replication. J Virol 81: 10280-10291, 2007. 11. Denison MR, Sims AC, Gibson CA, Lu XT: Processing of the MHV-A59 gene 1 polyprotein by the 3C-like proteinase. Adv Exp Med Biol 440: 121-127, 1998. 12. Gadlage MJ, Sparks JS, Beachboard DC, Cox RG, Doyle JD, Stobart CC, Denison MR: Murine hepatitis virus nonstructural protein 4 regulates virus-induced membrane modifications and replication complex function. J Virol 84: 280-290, 2010. 13. Groneberg DA, Poutanen SM, Low DE, Lode H, Welte T, Zabel P: Treatment and vaccines for severe acute respiratory syndrome. Lancet Infect Dis 5: 147-155, 2005. 14. Haagmans BL, Osterhaus AD: Coronaviruses and their therapy. Antiviral Res 71: 397-403, 2006. 15. Harcourt BH, Jukneliene D, Kanjanahaluethai A, Bechill J, Severson KM, Smith CM, Rota PA, Baker SC: Identification of severe acute respiratory syndrome coronavirus replicase products and characterization of papain-like protease activity. J Virol 78: 13600-13612, 2004. 16. Imbert I, Guillemot JC, Bourhis JM, Bussetta C, Coutard B, Egloff MP, Ferron F, Gorbalenya AE, Canard B: A second, non-canonical RNA-dependent RNA polymerase in SARS coronavirus. EMBO J 25: 4933-4942, 2006. 17. Ivanov KA, Hertzig T, Rozanov M, Bayer S, Thiel V, Gorbalenya AE, Ziebuhr J: Major genetic marker of nidoviruses encodes a replicative endoribonuclease. Proc Natl Acad Sci U S A 101: 12694-12699, 2004. 18. Keyaerts E, Vijgen L, Maes P, Neyts J, Van Ranst M: In vitro inhibition of severe acute respiratory syndrome coronavirus by chloroquine. Biochem Biophys Res Commun 323: 264-268, 2004. 19. Kim HY, Eo EY, Park H, Kim YC, Park S, Shin HJ, Kim K: Medicinal herbal extracts of Sophorae radix, Acanthopanacis cortex, Sanguisorbae radix and Torilis fructus inhibit coronavirus replication in vitro. Antivir Ther 15: 697-709, 2010. 20. Kim HY, Shin HS, Park H, Kim YC, Yun YG, Park S, Shin HJ, Kim K: In vitro inhibition of coronavirus replications by the traditionally used medicinal herbal extracts, Cimicifuga rhizoma, Meliae cortex, Coptidis rhizoma, and Phellodendron cortex. J Clin Virol 41: 122-128, 2008. 21. Kim JC, Spence RA, Currier PF, Lu X, Denison MR: Coronavirus protein processing and RNA synthesis is inhibited by the cysteine proteinase inhibitor E64d. Virology 208: 1-8, 1995. 22. Lai MM: Coronavirus: organization, replication and expression of genome. Annu Rev Microbiol 44: 303-333, 1990. 23. Madan V, Garcia Mde J, Sanz MA, Carrasco L: Viroporin activity of murine hepatitis virus E protein. FEBS Lett 579: 3607-3612, 2005. 24. Masters PS: The molecular biology of coronaviruses. Adv Virus Res 66: 193-292, 2006. 25. Perlman S, Netland J: Coronaviruses post-SARS: update on replication and pathogenesis. Nat Rev Microbiol 7: 439-450, 2009. 26. Sawicki D, Wang T, Sawicki S: The RNA structures engaged in replication and transcription of the A59 strain of mouse hepatitis virus. J Gen Virol 82: 385-396, 2001. 27. Sawicki SG, Sawicki DL: Coronavirus minus-strand RNA synthesis and effect of cycloheximide on coronavirus RNA synthesis. J Virol 57: 328-334, 1986. 28. Sawicki SG, Sawicki DL, Younker D, Meyer Y, Thiel V, Stokes H, Siddell SG: Functional and genetic analysis of coronavirus replicase-transcriptase proteins. PLoS Pathog 1: e39, 2005. 29. Schiller JJ, Kanjanahaluethai A, Baker SC: Processing of the coronavirus MHVJHM polymerase polyprotein: identification of precursors and proteolytic products spanning 400 kilodaltons of ORF1a. Virology 242: 288-302, 1998. 30. Stadler K, Masignani V, Eickmann M, Becker S, Abrignani S, Klenk HD, Rappuoli R: SARS--beginning to understand a new virus. Nat Rev Microbiol 1: 209-218, 2003. 31. Stern DF, Sefton BM: Coronavirus proteins: structure and function of the oligosaccharides of the avian infectious bronchitis virus glycoproteins. J Virol 44: 804-812, 1982. 32. Stockman LJ, Bellamy R, Garner P: SARS: systematic review of treatment effects. PLoS Med 3: e343, 2006. 33. Stohlman SA, Lai MM: Phosphoproteins of murine hepatitis viruses. J Virol 32: 672-675, 1979. 34. Tanner JA, Zheng BJ, Zhou J, Watt RM, Jiang JQ, Wong KL, Lin YP, Lu LY, He ML, Kung HF, Kesel AJ, Huang JD: The adamantane-derived bananins are potent inhibitors of the helicase activities and replication of SARS coronavirus. Chem Biol 12: 303-311, 2005. 35. te Velthuis AJ, van den Worm SH, Sims AC, Baric RS, Snijder EJ, van Hemert MJ: Zn(2+) inhibits coronavirus and arterivirus RNA polymerase activity in vitro and zinc ionophores block the replication of these viruses in cell culture. PLoS Pathog 6: e1001176, 2010. 36. Tooze J, Tooze S, Warren G: Replication of coronavirus MHV-A59 in sac- cells: determination of the first site of budding of progeny virions. Eur J Cell Biol 33: 281-293, 1984. 37. Weiss SR, Navas-Martin S: Coronavirus pathogenesis and the emerging pathogen severe acute respiratory syndrome coronavirus. Microbiol Mol Biol Rev 69: 635-664, 2005. 38. Wilson L, McKinlay C, Gage P, Ewart G: SARS coronavirus E protein forms cationselective ion channels. Virology 330: 322-331, 2004. 39. Wu CJ, Jan JT, Chen CM, Hsieh HP, Hwang DR, Liu HW, Liu CY, Huang HW, Chen SC, Hong CF, Lin RK, Chao YS, Hsu JT: Inhibition of severe acute respiratory syndrome coronavirus replication by niclosamide. Antimicrob Agents Chemother 48: 2693-2696, 2004. 40. Yu GY, Lai MM: The ubiquitin-proteasome system facilitates the transfer of murine coronavirus from endosome to cytoplasm during virus entry. J Virol 79: 644-648, 2005. 41. Zhai Y, Sun F, Li X, Pang H, Xu X, Bartlam M, Rao Z: Insights into SARS-CoV transcription and replication from the structure of the nsp7-nsp8 hexadecamer. Nat Struct Mol Biol 12: 980-986, 2005. 출처: https://www.ndsl.kr/ndsl/search/detail/article/articleSearchResultDetail.do?cn=DIKO0012485851 |